4-1.La multiplication des virus à ADN
4-2. La multiplication des virus à ARN
5. L’ASSEMBLAGE ET LA MATURATION
6. LA LIBERATION DES VIRUS
La multiplication virale est un phénomène complexe au cours duquel le virus va détourner la machinerie cellulaire à son profit. En effet, du fait de leur simplicité extrême, les virus ne peuvent pas se multiplier, du moins se multiplier par eux-mêmes. Connaître et bien comprendre les différentes étapes du cycle de multiplication virale est un objectif majeur pour le développement de molécules antivirales. Certaines étapes sont spécifiques du virus et constitue une cible idéale pour une molécule antivirale.
La multiplication d'un virus consiste en l'introduction du génome viral dans une cellule et c'est elle qui va fabriquer de nouveaux virus selon un procédé de biosynthèse que l'on appelle réplication.
Le temps du cycle viral peut varier d’un virus à l’autre en fonction de la taille du génome et de la complexité du cycle viral (4 à 8 heures pour le poliovirus, plus de 40 heures pour les Herpesviridae).
LA MULTIPLICATION D'UN VIRUS COMPORTE SIX ETAPES :
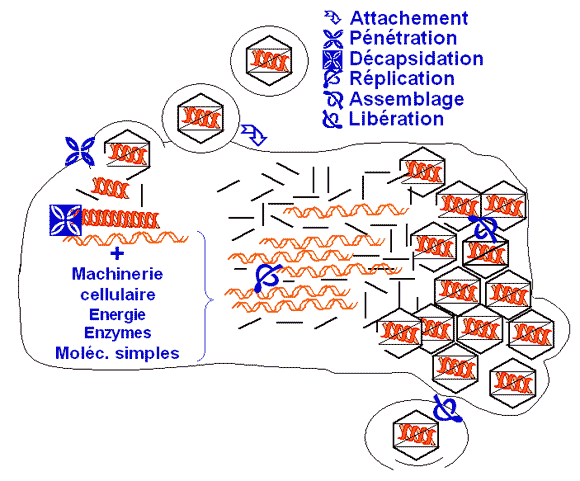
1. L'ATTACHEMENT
La première étape est l'entrée en contact du virus et de la cellule. C'est l'attachement de la surface virale sur la surface cellulaire. Cet attachement se fait par une structure de la capside pour les virus nus, par des glycoprotéines d’enveloppe pour les virus enveloppés. Ces protéines ou glycoprotéines s’attachent à des récepteurs situés sur la membrane cytoplasmique de la cellule hôte.
Ce besoin de récepteurs cellulaires de la membrane cytoplasmique pour les virus explique qu'un virus donné ne peut infecter qu'un nombre restreint d'espèces animales (tropisme d’hôte) avec des tropismes tissulaires et cellulaires précis. La sensibilité d’une cellule pour un virus définit sa capacité à pouvoir être infectée par un virus donné. L’ensemble des cellules sensibles à un virus définit son spectre d’hôtes. Ce dernier est variable selon les virus, certains peuvent infecter de nombreuses cellules, d’autres sont spécifiques d’un type cellulaire pour un animal donné.
Ainsi, les poliovirus n'infectent que l'homme et, expérimentalement, les singes supérieurs, mais pas les oiseaux, ni les poulets car les poliovirus ne trouvent de récepteurs pour leur attachement que sur les cellules de primates et non sur les cellules de poulet.
Les virus de l'immunodéficience humaine (HIV) infectent principalement les lymphocytes T CD4+ car leur enveloppe peut s'attacher sur la molécule CD4, récepteur spécifique de ces virus. La structure d'attachement de l'HIV est la glycoprotéine de surface de l'enveloppe, la gp120 (glycoprotéine de 120 000 daltons, 120 kDa de poids moléculaire).
Les virus de la grippe infectent principalement les cellules de l’arbre respiratoire car leur enveloppe se fixe sur les acides sialiques (récepteurs présents à la surface de ces cellules).
2. LA PÉNÉTRATION
On distingue trois mécanismes permettant l’entrée du virus à l'intérieur de la cellule, le plus souvent par microphagocytose pour les virus nus (ex : poliovirus) et, pour les virus enveloppés, par fusion de l'enveloppe virale et de la membrane cytoplasmique en une membrane unique, fusion suivie de lyse, par formation d'un pore (trou) qui s'élargit et laisse passer la capside dans le cytoplasme (ex : virus VIH). Un troisième mécanisme associe l’endocytose et la fusion. Après attachement sur son récepteur cellulaire, le virus est internalisé au sein d’une vésicule ou endosome. Une acidification du virus au sein de l’endosome est induite par une pompe à protons (ex : protéine M2 pour le virus influenza type A). Cela induit une modification conformationelle des protéines d’enveloppe virale libérant des régions protéiques hydrophobes habituellement cachées qui réagissent avec les lipides membranaires de l’endosome. Il s’ensuit une fusion des membranes et une libération de la capside virale dans le cytoplasme de la cellule. Ce mécanisme concerne de nombreux virus enveloppés (Orthomyxoviridae, Rhabdoviridae..).
3. LA DÉCAPSIDATION
Les structures virales vont ensuite être dégradées, à l'exception du génome qui, débarrassé de la capside, se trouve libéré. Il est nécessaire que la capside soit détruite pour que le génome, décortiqué, puisse fonctionner, livrer son information génétique à la machinerie cellulaire. En général cette étape se fait à l’aide de décapsidases cellulaires exception faite du poxvirus qui possède sa propre décapsidase. Après ces étapes d'initiation de l'infection prend place la phase de réplication et d'expression du génome viral.
4. LA RÉPLICATION
Le génome viral doit être transcrit, traduit et répliqué. Pour cela, le génome viral libéré prend la direction des synthèses, dans la cellule. Il se substitue en totalité ou en partie au génome cellulaire qui jusqu'alors organisait les synthèses cellulaires. Désormais dirigée par le génome viral, la cellule va détourner la machinerie cellulaire au profit du virus et va ainsi produire des virus entrainant dans certains cas une inhibition des synthèses d’ARN et d’ADN cellulaires.
Plus précisément, la cellule va faire des copies, (répliques) du génome viral, des répliques de protéines virales, protéines de capside et glycoprotéines d’enveloppe.
La stratégie de multiplication est dépendante de la nature et de la structure du matériel génétique : ADN ou ARN, génome bicaténaire ou monocaténaire, segmenté ou non, circulaire ou linéaire. Selon le type de virus la réplication sera plus ou moins complexe. Seuls les virus à ADN dont la réplication est intranucléaire peuvent utiliser les enzymes cellulaires pour la transcription. Les autres virus doivent posséder leurs propres enzymes (ex : poxvirus qui ont une réplication cytoplasmique, virus à ARN).
4-1. La multiplication des virus à ADN
Les virus à ADN double brin suivent des mécanismes de multiplication qui se rapprochent de ceux observés pour les gènes cellulaires. Le cycle viral peut être divisé en 2 phases sauf pour les Herpesviridae chez lesquels on distingue 3 phases :
- une phase précoce où une petite partie du génome est transcrite grâce à une ARN polymérase-ADN dépendante cellulaire. Les ARN messagers précoces migrent dans le cytoplasme cellulaire pour être traduits par les ribosomes de la cellule en protéines régulatrices non structurales ou en enzymes impliquées dans la synthèse de l’ADN.
Il y a ensuite réplication de l’ADN viral par l’ADN polymérase cellulaire ou virale aboutissant en un grand nombre de copies d’ADN viral. - une phase tardive où les ADN néoformés vont servir de matrices pour une deuxième transcription aboutissant à la formation d’ARN messagers tardifs qui après traduction vont former des protéines de structure (capside, enveloppe).
Quelques exemples :
Les Papillomaviridae, Polyomaviridae, Adenoviridae et Herpesviridae se multiplient dans le noyau. Ils interragissent largement avec les composants cellulaires les détournant pour assurer leur propre transcription virale.
Exemple du cycle de multiplication des Herpesviridae :
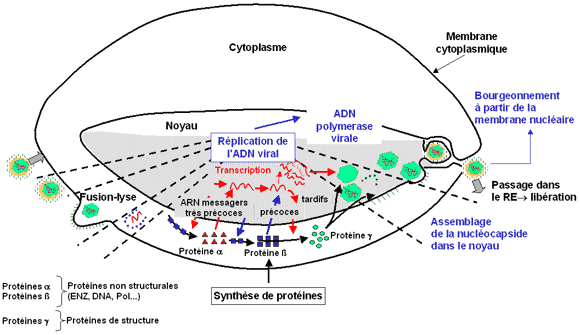
Les protéines virales sont synthétisées en trois phases : très précoce (protéines a), précoce (protéines b) et tardive (protéines g). Grâce à la protéine virale VP16 amenée par le virus dans la cellule, la transcription des gènes très précoces est initiée. Les protéines a synthétisées ont des fonctions de contrôle sur la transcription des gènes précoces et tardifs alors qu’elles inhibent par un phénomène d’autorégulation la transcription des gènes très précoces. Les protéines précoces b sont des enzymes impliquées dans la réplication de l’ADN viral. Elles activent également la transcription des gènes tardifs aboutissant à la synthèse de protéines structurales g.
Par contre, les Poxviridae se multiplient exclusivement dans le cytoplasme. Ils possèdent donc la plupart des facteurs nécessaires à leur réplication et sont indépendants de la machinerie cellulaire.
4-2. La multiplication des virus à ARN
Le génome des virus à ARN peut prendre différentes configurations : segmenté ou non, bicaténaire ou monocaténaire, de polarité positive ou négative.
Suivant les virus, l'élaboration des messagers viraux ou transcription est une opération plus ou moins complexe. Pour les virus à ARN de polarité positive tels que les poliovirus, tout est simple : le génome est un ARN qui sert tel quel de messager; il est immédiatement traduit par les ribosomes cellulaires en protéines de capside (et enzymes viro-induites). Pour les poliovirus, il n'y a pas de transcription. Par contre pour les virus à ARN de polarité négative tels que les orthomyxovirus ou les rhabdovirus, il faut passer par un ARN messager de polarité positive. Cette étape de transcription sera réalisée par une enzyme virale.
De même, la réplication du génome des virus à ARN nécessite l’apport d’enzymes spécifiques : ARN polymérase-ARN dépendante virale qui n’existe pas dans la machinerie cellulaire.
En effet dans la cellule normale, une telle opération et une telle enzyme n'ont pas de raison d'être et n'existent pas : les RNA cellulaires, qu'il s'agisse des RNA messagers, ribosomiques ou de transfert, sont synthétisés par des RNA polymérases DNA-dépendantes, travaillant sur une matrice de DNA, le génome cellulaire. Donc, pour se multiplier dans une cellule, un poliovirus et d’une façon générale tous les virus à RNA doit faire fabriquer à la cellule infectée une RNA réplicase, enzyme nouvelle, viro-induite, absente de la cellule normale, inutile au fonctionnement normal de la cellule, mais nécessaire à la multiplication virale. La transcriptase inverse (TI) ou rétrotranscriptase (RT) des rétrovirus est également une enzyme viro-induite.
A. Virus à ARN simple brin de polarité positive :
Exemple du cycle de multiplication du poliovirus :
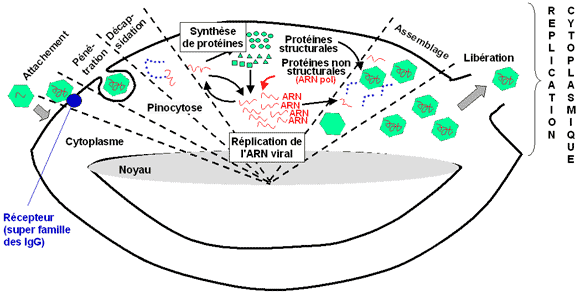
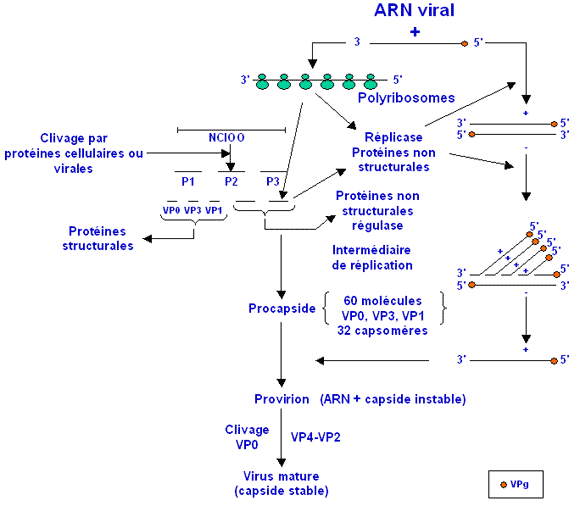
Le génome de polarité positive est traduit directement en une grande polyprotéine qui est ensuite clivée pour donner naissance à trois protéines P1, P2 et P3. La maturation de ces protéines virales fait intervenir plusieurs clivages en cascade. La région P1 contient l’information génétique codant pour les protéines de capside. Les régions P2 et P3 codent pour des protéines non structurales dont l’ARN polymérase-ARN dépendante ou réplicase. La réplicase va synthétiser un brin (-) pour aboutir à la forme réplicative constituée d’un brin (-) et d’un brin (+) appariés en double hélice. Le brin (-) va servir à la synthèse de nouveaux brins (+) toujours grâce à la réplicase dans un complexe appelé « intermédiaire de réplication ». Ces nouveaux brins (+) vont être encapsidés afin de former de nouveaux virions.
B. Virus à ARN simple brin de polarité négative :
Ils comprennent des virus à génome segmenté (Orthomyxoviridae, Bunyaviridae) ou non segmenté (Bornaviridae, Filoviridae, Paramyxoviridae et Rhabdoviridae). Ces génomes de polarité négative ne sont pas infectieux. L’infection nécessite une ARN polymérase associée au virion pour la transcription en ARN messager.
Exemple du cycle de multiplication des rhabdovirus :
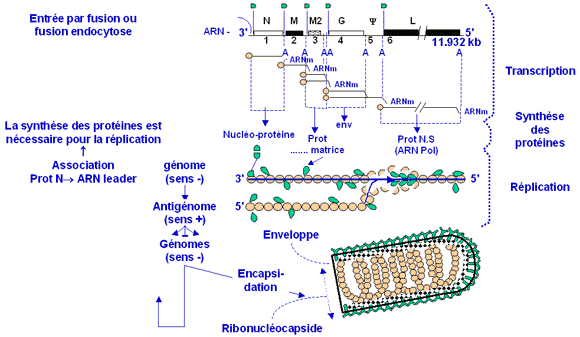
Il s’agit d’un virus non segmenté. Le génome est constitué de gènes séparés par des séquences intergéniques situées entre chaque gène. Ces séquences contiennent un signal de polyadénylation, de terminaison et d’initiation pour la transcription du gène suivant. La transcription des gènes est réalisée par l’ARN polymérase associée au virion. La quantité de messagers synthétisés varie selon la localisation des gènes (diminution de 3’ en 5’). Les ARN messagers sont traduits en protéines. Lorsque la concentration de nucléoprotéines (protéine N) atteint un certain seuil, celles ci se fixent sur l’ARN(+) nouvellement synthétisé et permettent à l’ARN polymérase de copier les jonctions intergéniques sans s’arrêter afin d’obtenir un ARN génomique entier.
C. La multiplication des virus utilisant la transcriptase inverse pour leur réplication
Une autre propriété spécifique de certains virus est la capacité de reproduire une molécule d’ADN à partir d’ARN (+) qu’il s’agisse d’ARN génomique pour les Retroviridae ou d’un produit de transcription pour les Hepadnaviridae (virus de l’hépatite B). Cette étape indispensable est rendue possible par la présence d’une enzyme virale, la transcriptase inverse (reverse transcriptase : RT).
Exemple du cycle de multiplication du virus de l’hépatite B :
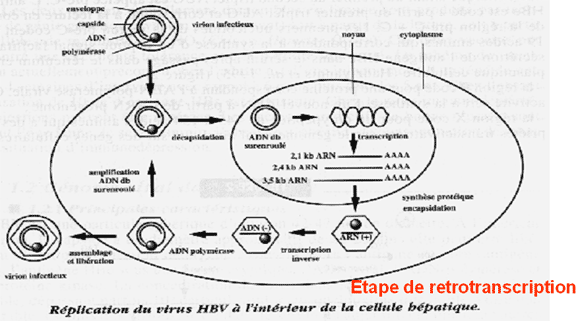
Contrairement aux autres virus à ADN infectant l’homme, le HBV se caractérise par un intermédiaire de réplication qui est une molécule d’ARN pré-génomique. C’est donc un virus à ADN (partiellement double brin, 1 brin négatif long, 1 brin positif court) qui se rapproche des rétrovirus par sa polymérase qui possède une activité de transcription inverse.
L’attachement du virus sur la cellule-cible (les hépatocytes) se ferait par interaction entre l’antigène préS1 côté virus et par l’albumine humaine polymérisé côté hépatocyte. La nature du récepteur de l'HBV n'est toutefois pas encore définie. Après migration des nucléocapsides dans le noyau, le génome viral y est libéré. Il acquiert une configuration circulaire superenroulée. Les ARN transcrits sont traduits en protéines dans le cytoplasme de l’hépatocyte (capside, protéines de surface, protéine X, polymérase virale). Un ARN viral prégénomique est également synthétisé puis encapsidé par 240 molécules de capside associée à la polymérase. La polymérase virale synthétise par son activité de transcription inverse le brin complémentaire négatif d’ADN (brin long). Après dégradation du brin d’ARN par son activité RNAseH, l’enzyme grâce à son activité ADN polymérase-ADN dépendante synthétise le brin + court pour aboutir à l’ADN circulaire partiellement double brin qui constitue le génome du virus.
Le HBV représente pour les virus à ADN un modèle en miroir des rétrovirus. Toutefois l’intégration de l’ADN viral dans le chromosome de la cellule n’est pas indispensable au cycle viral, elle peut cependant survenir au cours de l’infection chronique.
Exemple du cycle de multiplication du virus de l’immunodéficience humaine (VIH) :
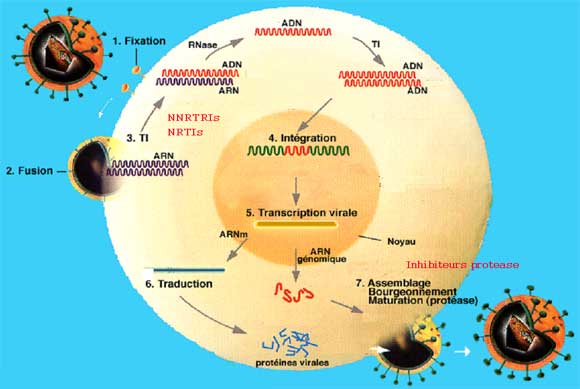
Une fois entré dans la cellule, l'ARN viral va être rétrotranscrit dans le cytoplasme en ADN par la transcriptase inverse virale (TI). La TI dégrade l'ARN viral puis copie l'ADN viral monocaténaire en ADN double brin qui passe dans le noyau de la cellule. Grâce à l'intégrase virale, l'ADN chromosomique est clivé et l'ADN viral s'intègre dans cet ADN chromosomique au sein du noyau de la cellule infectée, sous le nom d'ADN proviral. Une fois intégré et si il y a activation de la cellule, l'ADN proviral est transcrit en ARN messagers et en ARN génomique par l'appareillage de transcription de la cellule. La traduction d'ARN messagers peu ou pas épissés donne naissance à des polyprotéines virales correspondant d'une part aux gènes gag et pol et d'autre part au gène env. La polyprotéine env va être clivée par une protéase cellulaire en deux protéines d'enveloppe qui seront glycosylées par les enzymes de la cellule. L'assemblage des protéines virales et de 2 molécules d'ARN viral se fait au niveau de la membrane cellulaire. Quant à la polyprotéine Gag-Pol, c'est au moment du bourgeonnement du virus hors de la cellule qu'elle va être clivée par la protéase virale pour donner les protéines constitutives internes du virus et ses 3 enzymes. Cette dernière étape s'appelle la maturation et est indispensable à la production de virus infectieux capables d'infecter d'autres cellules.
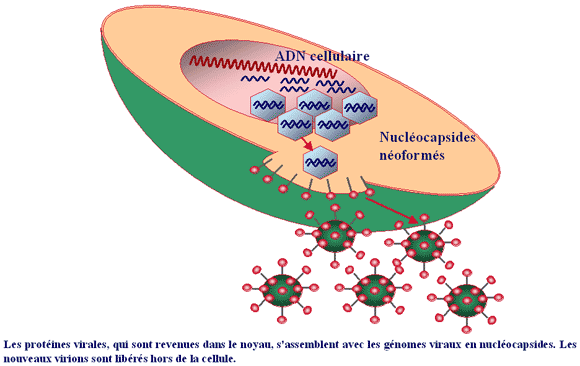
Les nouveaux génomes fabriqués par la cellule s'entourent de nouvelles protéines virales fabriquées par la cellule. C'est l'encapsidation des génomes qui aboutit à la formation de nouveaux virus. Les mécanimes peuvent être simples avec auto-assemblage des protéines de capside et encapsidation du génome. Ils peuvent être plus complexes avec l’intervention de protéines virales spécifiques.
6.LA LIBÉRATION DES VIRUS
Ces nouveaux virus sortent de la cellule par éclatement pour les virus nus, par bourgeonnement pour les virus enveloppés. C'est lors du bourgeonnement que les virus à enveloppe constituent leur enveloppe qui est une bicouche lipidique cellulaire hérissée de spicules glycoprotéiques. Certains virus comme les Herpesvirus s’entourent d’une enveloppe provenant de la membrane nucléaire de la cellule infectée, d’autres comme les rétrovirus s’entourent d’une enveloppe provenant de la membrane cytoplasmique de la cellule. Une cellule produit de l’ordre de 100 à 1000 virus.
|
|
|
|
|
