Toutes les infections virales ne nécessitent pas l'intervention du laboratoire.
Si le contexte clinique ou la gravité des symptômes le justifient, le diagnostic virologique fait appel soit aux méthodes indirectes, soit aux méthodes directes. Parmi ces dernières, le diagnostic moléculaire occupe une place de plus en plus importante.
|
Structure virale détectée
|
Méthode
|
Quelques applications courantes
|
| Diagnostic « indirect » | ||
| Anticorps anti-viraux | sérologies virales | presque tous les virus |
| Diagnostic « direct » | ||
| Particules virales complètes | culture virale | HHV, VZV, Entérovirus |
| Protéines virales | diagnostic direct « rapide » | Rotavirus, Adenovirus, RSV, Grippe, Virus Parainfluenza, HHV, VZV, CMV |
| Acides nucléiques viraux | biologie moléculaire | virus neurotropes (HHV-1…) HIV, HCV, HBV |
2 . Diagnostic indirect : sérologies virales
Pour le diagnostic d’une infection en cours, il est important d'analyser deux prélèvements consécutifs à 10-15 jours d’intervalle, pour observer une modification significative du taux d’anticorps. La présence d'IgM signe le plus souvent une infection récente ; les IgG persistent très longtemps.
Lorsqu’une personne est trouvée positive en sérologie pour la première fois pour le HIV et le HCV, il est légalement obligatoire de vérifier ce résultat sur un second prélèvement (sérologie et western blot).
2. 1. Prélèvements :
- Sérum principalement, acheminé au laboratoire à température ambiante.
- Autres liquides biologiques : liquide céphalo-rachidien (LCR), liquide amniotique, liquide pleural, liquide de lavage broncho-alvéolaire (LBA) ... Dans ce cas, une adaptation « artisanale » de la technique sérologique est souvent nécessaire, ce qui est peu satisfaisant du point de vue de l'assurance qualité.
| Prélèvements de sang en tube sec et tube gélosé pour la sérologie : le sang est centrifugé et des aliquots de sérum sont utilisés pour réaliser les sérologies prescrites. |
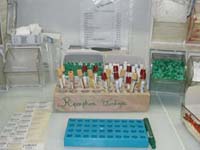 |
2.2. Techniques :
- Méthodes ELISA (Enzyme Linked Immunosorbent Assay) le plus souvent
- Autres réactions sérologiques, plus « traditionnelles », comme la réaction de fixation du complément
- Tests rapides (sensibilité variable, attention !)
- Tests de confirmation HIV (western blot) et HVC (immunoblot) : les différentes protéines du virus sont présentes, séparément, sur la membrane qui sert de support de réaction. Ces tests permettent de préciser contre quelles protéines virales sont dirigés les anticorps.
2.3. Principe de la réaction ELISA (enzyme linked immunosorbent assay) :
|
 |
Appareils de distribution de sérums et de gestion des méthodes ELISA : les sérums sont reconnus par lecture d'un code-barres, puis leur distribution et la réaction ELISA sont réalisées automatiquement, en connection avec le système informatique du laboratoire. Les résultats sont "validés"et interprétés individuellement par le technicien puis le biologiste, en fonction des valeurs des différents témoins et des renseignements cliniques.
 |
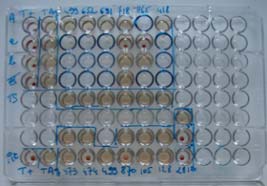
| Aspect d’une plaque de réaction ELISA en fin de manipulation : les puits fortement colorés correspondent à des sérums positifs pour la réaction ELISA. La lecture automatisée des densités optiques est réalisée sur un spectrophotomètre. |
 |
| Aspect d’une plaque de réaction de fixation du complément en fin de manipulation : dans les puits positifs, les globules rouges sont agglutinés les uns aux autres et forment un précipité au fond du puits. |
 |
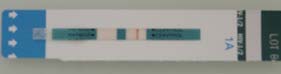
| Test sérologique HIV-1 et –2 rapide : ce type de test est particulièrement utile dans le cas d'un accident d'exposition au sang. Si le sérum du patient source est positif, un traitement anti-rétroviral est mis en route très rapidement chez le patient exposé |
 |
| Bandelettes de western blot en fin de réaction : une bande colorée apparait lorsque le sérum testé contient des anticorps reconnaissant l'antigène adsorbé à cet endroit-là sur la bandelette. |  |
- Indication : Mise en évidence d’un contact plus ou moins récent avec un virus.
- Avantages :
· Automatisation
· Délai d’obtention du résultat : le jour même (4 heures) ou le lendemain
· Bonne sensibilité, excellente spécificité (ELISA)
- Limites :
· Sensibilité moindre chez certains patients : nourrissons et personnes immunodéprimées.
· Interprétation délicate parfois. Exemple : pour tous les herpesviridae (HHV, CMV, EBV …) les IgM peuvent être présentes ou non lors des réactivations.
- Coûts indicatifs :
· sérologie HHV IgG et IgM : B70 + B70
· sérologie HVC : B70
· sérologie HIV (2 méthodes ELISA) : B70
· western blot HIV : B180
3. Diagnostic direct
3.1. Prélèvements :
La qualité du prélèvement conditionne le résultat. Il existe trois situations :
- Prélèvements ne nécessitant pas de milieu de transport : amener au laboratoire le prélèvement
· dans un tube sec stérile : selles, urines, LCR, prélèvements liquides divers
· dans un tube contenant de l'EDTA : sang total pour séparation au laboratoire des cellules mononucléées sanguines (recherche de CMV, EBV,HIV…)
- Prélèvements effectués avec un écouvillon stérile immergé dans un milieu de transport fourni par le laboratoire : gorge, vésicules, conjonctives
Dans ces deux cas,
· acheminer dans les 4 heures au laboratoire (pour préserver l’antigénicité, l’infectiosité ou la qualité des acides nucléiques),
· à 4°C en entourant le tube de prélèvement d'un sac de glaçons (pour éviter la prolifération bactérienne)
· cas particuliers :
- le sang total, à température ambiante
- pour les examens de Biologie Moléculaire : tube EDTA+ + + ( pas d’héparine, inhibiteur de la PCR)
- Frottis sur lame (l'écouvillon est exprimé sur la lame de verre par le préleveur) : acheminer à sec, à température ambiante
| Exemples de prélèvements : liquides céphalo-rachidiens, liquide de lavage broncho-alvéolaire, selles, un écouvillon stérile et un milieu de transport liquide |
 |
3.2. Les méthodes de diagnostic direct « rapide »
Ces méthodes permettent d'obtenir un résultat en quelques heures.
- Techniques :
· Immunomarquage spécifique d'un virus, sur lame de verre en 1 ou plusieurs « spots » (avec un anticorps dirigé contre une protéine virale), à partir d’un écouvillon ou de cellules mononucléées sanguines isolées au laboratoire. Préciser le virus recherché : un seul immunomarquage possible par « spot ».
· Autres possibilités : agglutination de particules de latex sensibilisées, méthode de type ELISA pour la détection automatisée des antigènes, trousses de détection immuno-enzymatique rapide d’antigènes…
- Indications :
Permettre une prise en charge spécifique et rapide du patient dans certaines circonstances :
· pre-partum (HHV),
· infections respiratoires (Grippe, Virus Respiratoire Syncytial, virus Parainfluenza, Adénovirus),
· diarrhées du nourrisson (Rotavirus, Adénovirus),
· recherche d’une infection à CMV infra-clinique chez un patient immunodéprimé (antigénémie pp65).
| Test d’agglutination pour la recherche de Rotavirus dans les selles : les selles, suspendues dans un tampon, sont mises en présence de particules de latex "sensibilisées" à l'antigène viral. Le résultat est négatif dans le cas illustré. Dans le cas contraire, des mesures d'hygiène strictes permettraient de limiter l'extension de l'épidémie de gastro-entérite parmi les nourrissons hospitalisés. |
 |
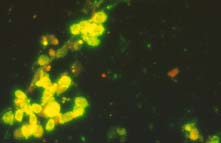
| Immunofluorescence spécifique du HHV sur un frottis vaginal : les cellules infectées présentent une fluorescence jaune-verte. Un accouchement par césarienne va être proposé à la patiente. |
 |
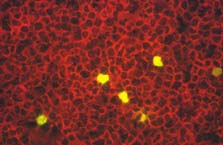
| Antigénémie pp65 du CMV positive : les leucocytes marqués par l'anticorps (fluorescence jaune-verte) contiennent la phosphoprotéine de 65kD du CMV. Le patient, greffé rénal, va bénéficier d'un traitement contre le CMV, même s'il ne présente pas encore de signe clinique (traitement pré-emptif). |
 |
- Avantage :
rapidité d’obtention du résultat : 20 minutes à 4 heures
- Limites :
· Manque de sensibilité + + + . Un résultat négatif n'exclut pas le diagnostic.
· Immunofluorescence : lecture au microscope fonction de l'expérience de l'observateur.
- Coût :
· immunofluorescence HHV sur prélèvement vaginal : B40
· recherche d’antigène rotavirus dans les selles : B40
· antigénémie pp65 CMV : B 100
3.3. Les méthodes de diagnostic direct par culture
Les virus sont des parasites obligatoires des cellules : ils sont cultivés dans des cellules eucaryotes entretenues au laboratoire, sous la forme de lignées dites continues ou semi-continues.
- Les différentes étapes du diagnostic par culture virale
1. Ensemencement d'une lignée cellulaire avec le prélèvement pathologique.
2. Examen direct quotidien: recherche quotidienne d'un effet cytopathique (ECP) au microscope optique inversé.
3. Si ECP : coloration des cellules, pélevées et étalées sur lame, à la recherche d'inclusions virales et d'altérations cytoplasmiques ou nucléaires, au microscope optique.
4. Immunomarquage spécifique du virus sur lame, avec lecture au microscope optique ou à fluorescence.
| Poste de travail pour les cultures virales : hotte à flux laminaire, matériel jetable et stérile. |
 |
| Observation de cultures cellulaires (tubes, boîtes multi-puits de plastique et flacons) au microscope optique inversé |
 |
|
Effet cytopathique évocateur d’une infection par HHV dans une culture de cellules MRC5 (fibroblastes embryonnaires humains) |
 |
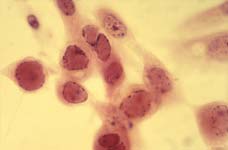
| Coloration cellulaire montrant les particularités de cet ECP : disparition des nucléoles, chromatine éclatée sur les bords de la membrane nucléaire, inclusions nucléaires fortement colorées. |
 |
| Par comparaison, ECP caractéristique d’une culture d’entérovirus : inclusions intra-cytoplasmiques et noyau hyperdense repoussé en périphérie de la cellule. |
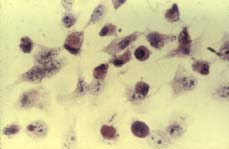 |
- Indications :
· Isoler un virus dans un prélèvement biologique.
· Prouver le caractère infectieux du virus.
· Caractériser le phénotype d’une souche virale (résistance aux anti-viraux par exemple).
- Avantages :
· Bonne sensibilité + + +. La culture demeure la référence à laquelle toutes les nouvelles méthodes sont comparées.
· Elle est indispensable pour disposer de souches, utilisées par exemple dans la mise au point des vaccins (ex : vaccin anti-grippal chaque année).
· Coût raisonnable.
- Limites :
· Délai usuel d’obtention du résultat : 48 heures à 10 jours.
· Subjectivité de la lecture : expérience de l'observateur +++.
· La culture est pratiquée dans peu de laboratoires de ville.
· Certains virus ne sont pas cultivables en dehors de laboratoires de recherche très spécialisés.
- Coût :
· culture HHV sur prélèvement génital : B150
· culture entérovirus dans les selles : B150
3.4. Le diagnosticdirect moléculaire
Pour simplifier, trois méthodes principales dominent la routine : l’hybridation moléculaire et ses variantes (hybridation avec amplification du signal ou bDNA, hybridation à la recherche de mutations du génome viral), l’amplification génique ou PCR, et le séquençage nucléotidique.
Les applications quantitatives deviennent courantes
|
 |
3.4.1. Hybridation avec amplification du signal
- Principe de l’ADN branché :
L’acide nucléique viral (prélèvement) est
· adsorbé sur un support grâce à des sondes de capture, puis
· détecté grâce à des sondes ramifiées sur lesquelles
· s’hybrident des sondes de révélation.
Des molécules luminescentes sont présentes sur les sondes de révélation; le signal luminescent est proportionnel au nombre de molécules d'acide nucléique viral adsorbées.
L'utilisation simultanée de nombreuses sondes ramifiées, complémentaires de différentes régions du génome viral cible, permet la détection de virus génétiquement très variables (HIV, HVC). Le grand nombre de molécules luminescentes en jeu permet une diminution du seuil de détection, sans risque de contamination (il n’y a pas amplification de l’acide nucléique, contrairement à une PCR).
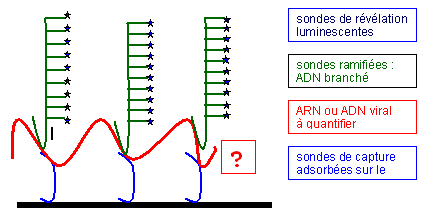
- Applications courantes : ARN HIV-1 , ARN HVC, ADN HVB
3.4.2. Hybridation sur membrane et puces
- Principe des différentes méthodes:
1) hybridation à des sondes moléculaires fixées sur un support et spécifiques soit d’un virus sauvage, soit d’un virus muté, puis
2) détection immuno-enzymatique.
Les sondes sont présentes sur des bandelettes, un peu à la manière des antigènes fixés sur les bandelettes d'immunoblots (ex : génotypage du HVC, mutations du HVB).
Les "puces" permettront elles aussi la détection d’acides nucléiques viraux mutés. Des sondes en grand nombre sont adsorbées sur des supports de silice, et représentent une sorte de combinaison des techniques informatiques et moléculaires. Une application sera rapidement disponible pour la détection de mutations du HIV associées à la résistance aux anti-rétroviraux.
|
Génotypage du HVC par hybridation sur membrane après PCR : les bandes sont interprétées à l’aide d’un calque fourni par le fabricant. Dans ce cas, le patient est porteur d'un HVC de génotype 1, de sous-type b, plus volontiers résistant au traitement anti-HVC. |
 |
3.4.3. Amplification génique
- Principe de la PCR :
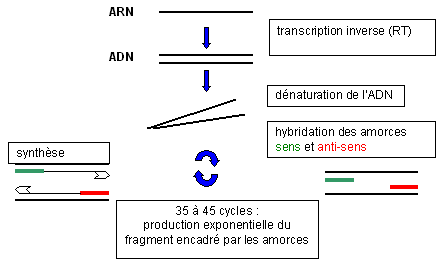
· A partir d’un prélèvement, l’ADN ou l’ARN sont extraits, selon différentes méthodes. Souvent, en routine, l’extraction est obtenue par adsorbtion des acides nucléiques (chargés négativement) sur des particules de silices chargées positivement, puis élution.
· L’amplification génique, ou PCR (polymerase chain reaction) permet le recopiage d’une portion limitée du génome viral, grâce à des amorces et à l’action d’une ADN polymérase.
· Les amorces sont conçues de telle sorte qu’elles s’hybrident au génome viral, l’une sur le brin sens, l’autre sur le brin anti-sens. On ne peut donc choisir des amorces que pour un virus dont on connaît la séquence nucléotidique ; de plus, les amorces doivent être spécifiques du virus, et ne s’hybrider à aucun autre génome que celui qui est recherché.
· L’ADN polymérase « s’accroche » aux deux amorces et synthétise de l’ADN complémentaire du génome viral qui lui sert de modèle ; la synthèse est possible grâce à l’apport dans le milieu réactionnel de dATP, dCTP, dGTP, dTTP. Chaque cycle de PCR comporte une étape de dénaturation de l’ADN, une étape d’hybridation des amorces, puis une étape de synthèse de l’ADN, étapes réalisées dans un thermocycleur par trois changements successifs de température (ex : 94°C, 60°C, 72°C).
· Pour amplifier l’ARN, une étape préliminaire de transcription inverse en ADN est indispensable avant la PCR ; elle est réalisée in vitro grâce à l’action d’une transcriptase inverse (d’origine aviaire ou murine). Tous les réactifs cités ci-dessus sont commercialisés.
· Après l’amplification génique, il est nécessaire de détecter les acides nucléiques amplifiés, soit par électrophorèse en gel d’agarose, soit plus volontiers par hybridation moléculaire post-PCR.
- Applications courantes : HVC, HVB, CMV, HHV, virus neurotropes, parvovirus B19, …
| Quatre types de thermocycleurs conventionnels : un thermocycleur est un bain-Marie à sec programmable (température et durée). |  |
gel d’agarose après PCR et electrophorèse. Le gel est photographié sous lumière UV. Les signaux positifs apparaissent sous l’aspect de bandes claires nettes (flèche). Le résultat n'est interprétable que s'il existe des témoins positifs (la réaction a fonctionné) et un témoin négatif (il n'existe pas de contamination).
 |
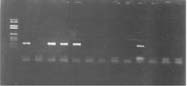 |
| Aspect d’une microplaque d’hybridation moléculaire post-PCR : les puits colorés correspondent aux signaux positifs. |
 |
3.4.4. Séquençage nucléotidique
La méthode de Sanger est utilisée pour le séquençage de routine. C'est une PCR, au cours de laquelle on ajoute des didéoxynucléotides (ddATP, ddCTP, ddGTP, ddTTP) marqués par un fluorochrome au mélange réactionnel habituel. La polymérase synthétise l'ADN en utilisant, au hasard, soit les déoxynucléotides, soit les didéoxynucléotides. Chaque fois qu'un didéoxynucléotide est incorporé dans la chaîne d'ADN, l'élongation de la chaîne s'arrête, car ces molécules ne permettent pas la liaison phospho-diester suivante. La réaction de séquence aboutit donc à la fabrication de fragments d'ADN de toutes les tailles possibles (par exemple, pour un fragment d'ADN recopié de 200 nucléotides, de 1 à 200). Les séquenceurs « automatiques » permettent la détection automatique du fluorochrome sur ces fragments d’ADN, par électrophorèse. Celle-ci est réalisée soit en gel vertical d’acrylamide, soit dans des capillaires de sicile remplis de polymère.
| Exemple de séquenceur automatique : les capillaires d’électrophorèse et le système de lecture sont situés dans l’appareil de gauche. L’interprétation se fait à l’aide d’un logiciel spécialisé, sur un PC directement connecté au séquenceur. |
 |
Analyse d’une réaction de séquence après lecture sur un séquenceur automatique : les deux brins d’ADN, sens et anti-sens, ont été séquencés (deux électrophorégrammes) et les séquences sont comparées entre elles et à une séquence de référence connue. Le « consensus » représente la séquence finale, obtenue après correction humaine des ambiguités non résolues par le séquenceur automatique.
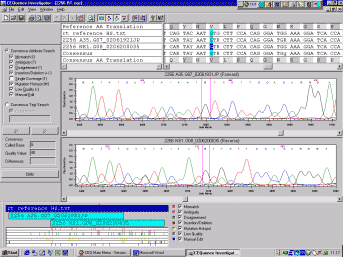
3.4.5. Quantification
· L’ADN branché est une méthode de quantification.
· PCR compétitive avec standard (ou étalon) interne
- Prinicpe de la Quantification par PCR compétitive :
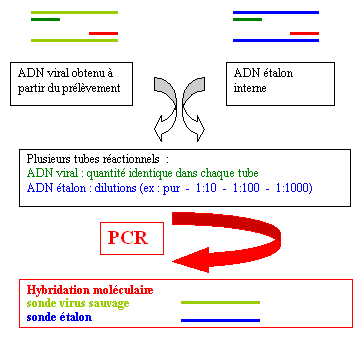
Zone s’équivalence : densité optique virus savage = densité optique étalon
Différentes trousses de PCR commercialisées sont fondées sur ce principe. Un étalon est introduit dans les prélèvements à analyser, avant l’extraction des acides nucléiques (étalon interne). Cet étalon est un acide nucléique dont les extrémités sont identiques à celles de la séquence du virus sauvage, ce qui permet d’utiliser le même couple d’amorces. Par contre, entre les extrémités auxquelles s’hybrident les amorces, la séquence nucléotidique diffère de celle du virus sauvage, ce qui permet de distinguer les deux produits de PCR, soit par électrophorèse (taille différente), soit par hybridation après PCR (sondes spécifiques différentes).
Chaque échantillon est testé dans plusieurs tubes. Dans chacun des tubes, le prélèvement est présent toujours en même quantité, mais l’étalon est distribué avec des concentrations différentes (dilutions de l’étalon). Au cours de la PCR, les amorces s’hybrident soit à la séquence sauvage (virus de l’échantillon), soit à l’étalon : il y a donc compétition (PCR compétitive). Les amorces s’hybrident plus volontiers à l’ADN présent en quantité supérieure : il est donc davantage amplifié. Dans un tube la même amplification est obtenue pour les deux matrices (zone d’équivalence) : la concentration d’étalon étant connue, cela permet de déduire la quantité de molécules dans l'échantillon.
· PCR « en temps réel »
Les méthodes de PCR en temps réel mesurent la quantité de molécules d’ADN fabriquées pendant le début de la phase exponentielle de la réaction de PCR. Schématiquement, l'ADN fabriqué pendant la PCR est quantifié par incorporation d’une molécule fluorescente ou hybridation de deux sondes spécifiques émettant une fluorescence. Ces thermocycleurs particuliers sont équipés pour la lecture continue (en temps réel) de la fluorescence émise.
|
Exemple d'appareil de PCR en temps réel : la PCR et la détection par hybridation sont réalisées en même temps dans le même tube.
|
 |
| Courbes de quantification de l’EBV dans le sang total par PCR en temps réel : les courbes du haut illustrent l'amplification de l'ADN des témoins positifs à différentes concentrations (courbe étalon externe). La courbe du bas représente la pente …. |
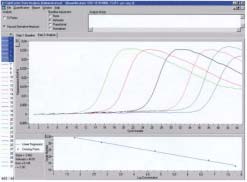 |
- Indications des méthodes de diagnostic moléculaire :
· Techniques qualitatives : montrer la présence d’un virus, en particulier lorsqu’il n’est pas cultivable (HVC), ou que le liquide biologique se prête mal à la culture (LCR).
· Techniques quantitatives : suivi des patients sous traitement anti-viral (HIV, HVC, CMV, HVB).
· Séquençage : détection de mutations nucléotidiques
- associées à la résistance aux anti-viraux (HIV, HVB),
- caractéristiques des génotypes des virus (HVC, HVB).
- Intérêts :
. Sensibilité remarquable
· Automatisation possible
- Limites :
· Risques de contamination pour les techniques de PCR.
· Absence de preuve du caractère infectieux du virus détecté.
· Caractérisation phénotypique de la souche impossible.
· Coût encore relativement élevé.
Ce cours a été préparé par Madame le Pr. M.E. Lafon (Laboratoire de Virologie, Faculté de Médecine, Université Victor Segalen , Bordeaux).
Pour en savoir plus : « Virologie médicale » A. Mammette, Collection AZAY, Presses Universitaires de Lyon. 2002, 80 Boulevard de la Croix Rousse, BP 4371, 69242 LYON 04
|
|
|
|
|
